|
16S rRNA扩增子测序
16S rRNA基因是对原核微生物进行系统发育和分类研究时最常用的分子标志物,广泛应用于微生物生态学研究中。近年来随着高通量测序技术及数据分析方法的不断进步,大量基于16S rRNA基因的研究使得微生物生态学得到了迅速发展。16S rRNA是原核微生物核糖体30S小亚基的组成部分,其rDNA序列包含10个保守区域和9个高变区域。相较于5S rRNA和23S rRNA基因序列,16S rRNA基因序列长度合适,其中保守区域在细菌间差异不大,高变区域序列具有原核微生物种间特异性的优点,因此被广泛应用于原核微生物的系统发育分析和物种的分类。罗宁生物16S rDNA测序采用Illumina测序技术的分析平台,具有高通量、高精度、极佳重现性和测序成本低等特点,目前已成为研究环境微生物中细菌或古菌的组成和多样性的主流方法。

16S rRNA基因序列组成及引物选择
引物信息

*覆盖度较传统V4区引物对更高的U515F-M/E806R-M引物对现已免费提供。如何选择引物请参阅16S引物选择说明。
技术路线

技术参数
| 测序平台 | Illumina Miseq / Hiseq 2500 |
|
|
|
| 测序深度 | 1-20万条Tags,Q30≥90% |
|
|
|
|
参考数据库 |
RDP, SILVA, Greengenes |
|
|
|
| 分析内容 | 序列拼接 | Tags过滤 | 去嵌合体 | OTU聚类 |
|
|
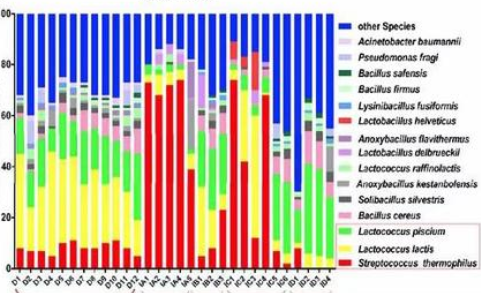
物种分类注释 | 物种丰度热图 |
进化树重构建 |
组间差异分析 |
|
|
Alpha多样性 | Beta多样性 | PCA/PCoA/NMDS | RDA/db-RDA/CCA |
|
|
Network分析 | Machine Learning | UniFrac距离箱形图分析 | |
|
|
PerMANOVA | Core Taxa | Mantel Test | PICRUSt |
|
|
null model | Plspm |
Niche Breadth |
VPA |
|
|
MRM | Response ratio | 时间序列模型 | 肠型分析 |
|
|
主成分回归 | 变异系数 | 培养基预测 |
|
| 项目周期 | 28-38个自然日 |
|
|
|
样品要求
1. 新鲜样本 (请使用干冰运送)
土壤、淤泥、沉积物, 大于2g。
建议使用试剂盒:Quick-DNA Soil Microbe Kits,DNeasy PowerSoil Kit
肠道样本, 大于0.5g
建议使用试剂盒:Quick-DNA Fecal Microbe Kits,QIAamp Fast DNA Stool Mini Kit
水体样本, 2-5L水样过滤后的滤膜
建议使用试剂盒:DNeasy PowerWater Kit
拭子样本, 大于2个
建议使用试剂盒:ZymoBIOMICS DNA Kits
2. 纯化核酸样本 (请使用干冰或冰袋运输)
纯化DNA样本,浓度大于5ng/μl(如使用NanoDrop测定浓度,样本浓度大于10ng/μl),总量大于150ng。核酸样本主带明显,无降解,无RNA或蛋白质污染。
PCR产物浓度大于25ng/μl,总量大于400ng,片段小于450bp。样本条带单一,无降解,无引物二聚体。